CRISPR/Cas9 : le génie génétique atteint une nouvelle dimension
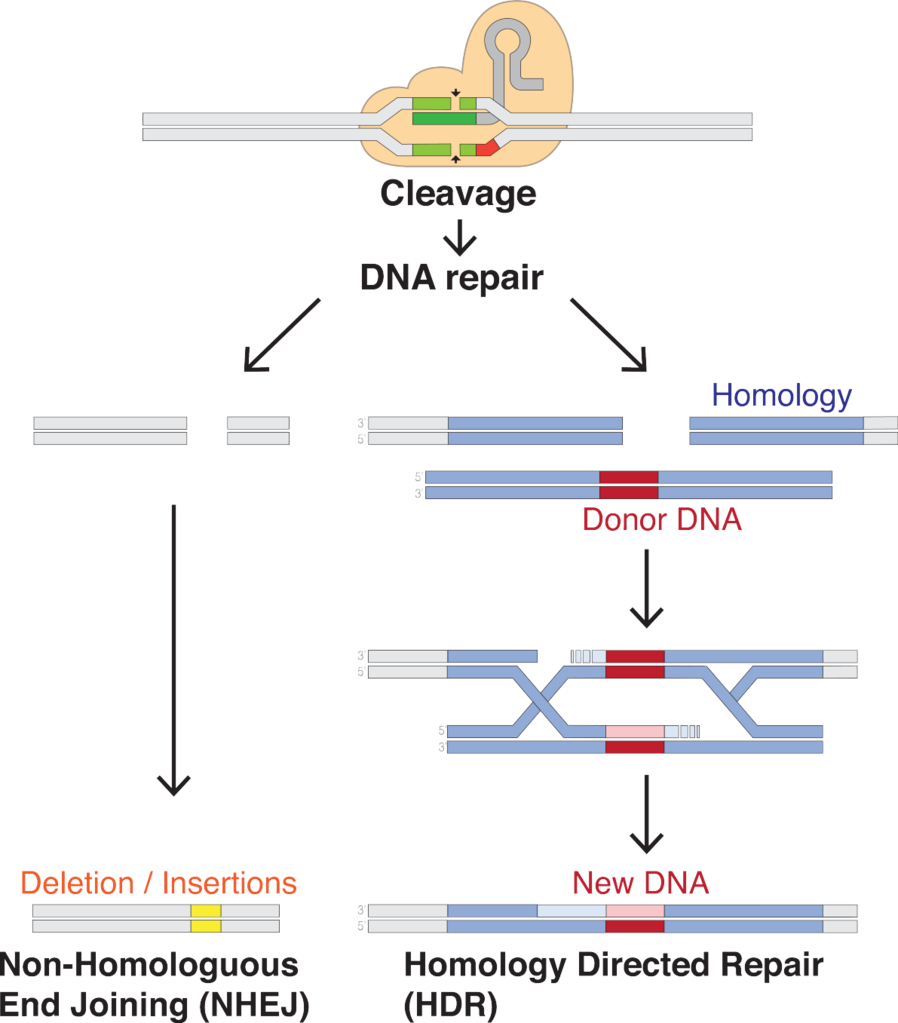
Dans le quotidien du laboratoire, les scientifiques sont régulièrement amenés à modifier l'ADN, par exemple pour étudier la fonction de certaines protéines. Il existe déjà diverses méthodes permettant ce que l'on appelle l'édition génomique, mais CRISPR/Cas9 est maintenant considérée comme la méthode de choix lorsqu'il s'agit de couper l'ADN à un endroit précis. La difficulté n'est pas de couper l'ADN - il existe pour cela des enzymes connues depuis longtemps - mais de le couper à un endroit précis. Le système de modification génomique a donc besoin, en plus d'un outil tranchant, d'un mécanisme de reconnaissance de la séquence cible d'ADN.
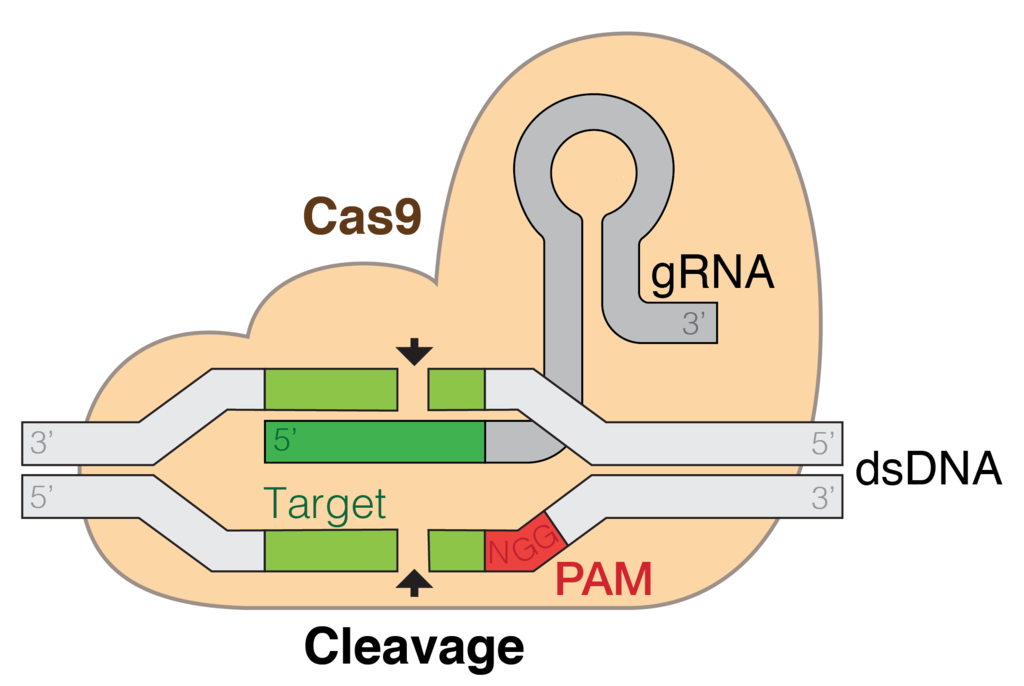
gRNA et Cas9 : reconnaître et couper
Les deux éléments qui constituent la base de la méthode CRISPR/Cas9 sont l'ARN guide (guide RNA ou gRNA, souvent appelé aussi single guide RNA, ARN guide simple) et la protéine Cas9 (CRISPR associated protein), la véritable « paire de ciseaux » génétique. Ces deux éléments forment un complexe. Le gRNA est synthétisé en laboratoire de manière à être complémentaire de la séquence d'ADN cible - il reconnaît donc la cible qui doit être coupée. Sa longueur est en général de 17 à 21 nucléotides, ce qui garantit que la séquence n'apparaît qu'une seule fois dans le génome et que l’ADN ne sera pas sectionné en dehors de la région souhaitée. Cependant, avant de pouvoir reconnaître et couper la séquence d’ADN, le complexe gRNA/Cas9 doit d'abord l'atteindre.
Motif PAM : sésame ouvre-toi
Les motifs PAM ne sont rien d'autre que de courtes séquences d'ADN qui sont reconnues par les protéines Cas. Dans le cas de Cas9, qui a été isolée à partir de la bactérie Streptococcus pyogenes, il s'agit de la séquence NGG, N pouvant être n'importe quel nucléotide. La protéine Cas9 se lie à un motif PAM et déroule l'hélice d'ADN à cet endroit - la séquence d'ADN est maintenant accessible et peut théoriquement être coupée.
Le fait que les motifs PAM ne comportent que quelques nucléotides est un avantage pour le génie génétique. Ces courtes séquences de nucléotides sont relativement fréquentes dans le génome, de sorte que l'on peut généralement trouver plusieurs PAM dans un même gène. La probabilité qu'un PAM se trouve à proximité de la séquence cible que l'on veut couper, est donc élevée. Pourtant, les motifs PAM ne se trouvent pas toujours exactement là où l'on souhaite couper l'ADN. En plus de Cas9, les scientifiques ont découvert de nombreuses autres protéines Cas, qui fonctionnent toutes de façon similaire mais ciblent des motifs PAM différents. Cela permet une modification du génome plus flexible et encore mieux ciblée.