Viele DNA-Sequenz-Wiederholungen – Viele Fragen der Wissenschaftler
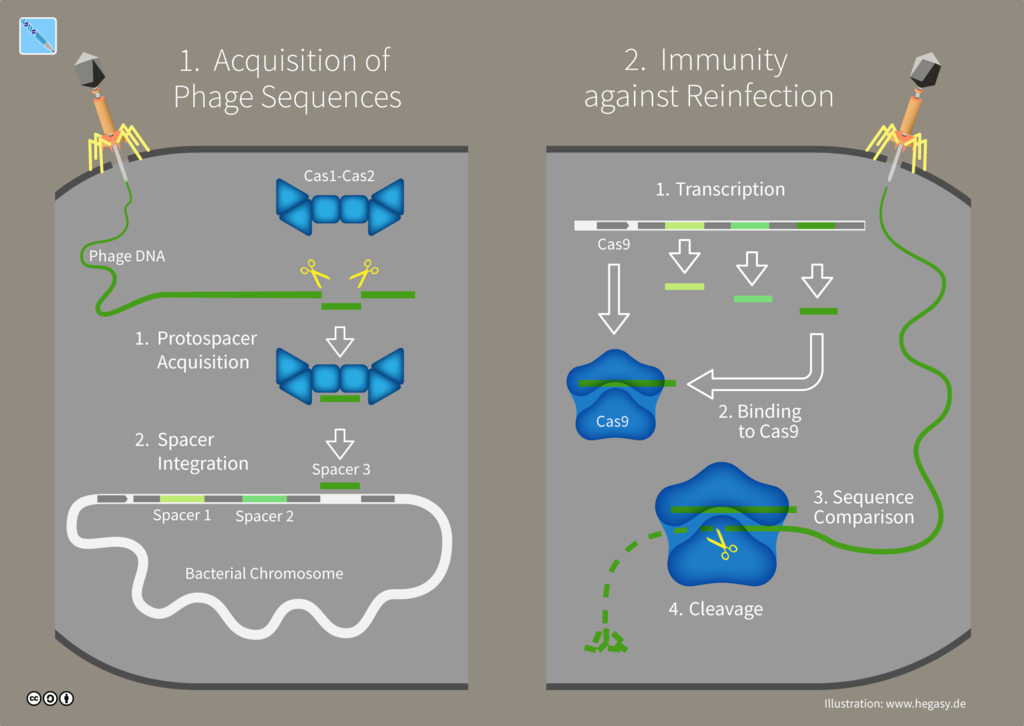
Nach der Entdeckung von Ishino in E. coli Bakterien im Jahre 1987, fanden Forscher wenige Jahre später auch in Mycobacterium tuberculosis, dem Erreger der menschlichen Tuberkulose, solche kurze, sich wiederholende DNA-Sequenzen. Bis zur Jahrtausendwende wurden dann von Forschern rund um den Globus immer mehr Bakterienarten gefunden, die ähnliche Eigenschaften aufwiesen. Im Jahre 2002 wurde erstmals die Abkürzung "CRISPR" (ausgesprochen Krisper) benutzt, um diese Sequenzen zu benennen. Das Akronym "CRISPR" steht für Clustered Regularly Interspaced Short Palindromic Repeats; zu Deutsch etwa kurze, palindromische Wiederholungen in regelmässigen Abständen.
Zwar wussten Wissenschaftler zu diesem Zeitpunkt bereits einiges über den Aufbau dieser DNA-Abschnitte, beispielsweise dass sie zwischen 23 und 47 Basenpaare lang sind und immer in einem Abstand von 21 bis 72 Basenpaaren vorkommen, aber ihre eigentliche Funktion war immer noch unklar. Im Jahr 2005 entdeckte man, dass die Sequenzen zwischen den CRISPR-Abschnitten, die sogenannten Spacer, identisch mit Abschnitten von Phagen-DNA sind. Erst im Jahre 2007 konnte dann ein Forscher-Team aus den USA zeigen, dass Bakterien diese Spacer-Abschnitte als Vorlage benutzen, um Phagen zu erkennen.