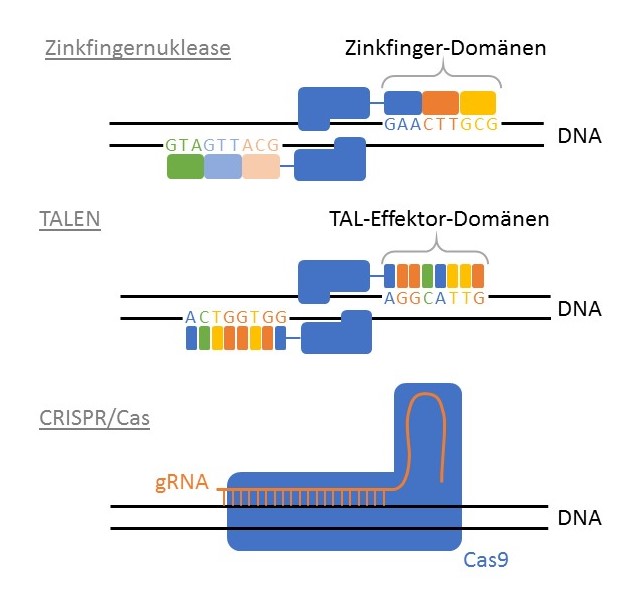
Zinkfingernukleasen – Dem Ziel einen Schritt näher
Zinkfingernukleasen sind künstlich hergestellte Restriktionsenzyme, die im Vergleich zu den vorher beschriebenen, gezielter arbeiten. Das Spezielle an diesen Enzymen ist, dass sie aus zwei Proteindomänen bestehen: der Zinkfingerdomäne und der Nukleasedomäne. Setzt man diese zwei Teile des Enzyms zusammen, ergibt sich auch der Name dieser Enzyme: Zinkfingernukleasen. Die Nukleasedomäne ist für das Schneiden der DNA zuständig, während die Zinkfingerdomäne so gebaut werden kann, dass sie spezifische DNA-Sequenzen erkennt. Meistens kommen mehrere Zinkfingermotive zusammen vor, um die Zinkfingerdomäne zu bilden. Jedes Zinkfingermotiv erkennt drei DNA-Basenpaare. Werden nun mehrere dieser Motive hintereinandergeschaltet, wird das Erkennen der DNA immer genauer. Eine Sequenz von drei Basenpaaren kommt in der DNA häufig vor, eine Sequenz von 6, 9, 12, 15 oder 18 Basenpaaren aber immer weniger häufig. Meistens werden maximal sechs Zinkfingermotive hintereinander eingefügt, da es technisch immer schwieriger wird noch grössere Zinkfingernukleasen zu bauen. Ausserdem beeinflussen die Motive ihre Erkennungssequenzen gegenseitig, weshalb es nicht ausreicht einfach die Erkennungssequenz von den einzelnen Motiven zu kennen, um eine grössere DNA Erkennungssequenz zu bauen. Weltweit können nur wenige Labors massgeschneiderte Zinkfingernukleasen herstellen und deren Herstellung kann Monate dauern und Tausende von Dollar kosten.
TALENs – Vereinfachte Herstellung
Der Aufbau der TALENs (transkriptionsaktivatorartige Effektornukleasen) ist sehr ähnlich zu dem der Zinkfingernukleasen. Auch hier besteht das Enzym aus zwei Untereinheiten, eine, welche die DNA schneidet und eine, welche die DNA-Sequenz erkennt, den TAL-Effektor. Eine TAL-Effektor-Domäne besteht meistens aus 34 Aminosäuren, von denen zwei variabel sind und bestimmen, welche DNA-Base erkannt wird. Werden viele TAL-Effektordomänen mit unterschiedlicher DNA-Basen-Spezifität hintereinandergeschaltet, können lange DNA-Sequenzen erkannt und gebunden werden. TALENs im Labor herzustellen hat sich als einfacher herausgestellt als Zinkfingernukleasen herzustellen.
CRISPR/Cas – Besser als alle anderen Methoden?
Der grosse Vorteil von CRISPR/Cas beruht auf der Einfachheit, mit der das System auf die jeweilige DNA-Zielsequenz angepasst werden kann. Im Gegensatz zu Zinkfingernukleasen und TALENs muss dafür kein neues Protein mit unterschiedlichen Motiven, sondern nur eine ca. 20 Basenpaar lange RNA synthetisiert werden, was 1-2 Tage dauert und nicht mehr als eine Pizza kostet. CRISPR/Cas ist somit eine Methode, die sich auch kleine Labors leisten können. Hinzu kommen die bisher unerreichte Effizienz und Spezifizität der Methode, die ein breites Spektrum an Anwendungen ermöglichen. Aber auch bei CRISPR/Cas sind technische Schwierigkeiten nicht ausgeschlossen (Lies dazu den Artikel "CRISPR/Cas: Fragen und Regeln"). Forschende entwickeln deshalb die Methode stetig weiter, um sie präziser zu machen und mehr Anwendungen zu ermöglichen.