CRISPR/Cas9: Gentechnik erreicht neue Dimensionen
Im Laboralltag sind Forschende regelmässig darauf angewiesen DNA zu verändern, um z. B. die Funktion bestimmter Proteine zu untersuchen. Es gibt bereits verschiedene Methoden, die eine sogenannte Genomeditierung ermöglichen, CRISPR/Cas9 aber gilt inzwischen als die Methode der Wahl, wenn es darum geht DNA an einer präzisen Stelle zu schneiden (warum das so ist, kannst du im Artikel "Genomeditierung vor dem CRISPR/Cas- Zeitalter" nachlesen). Die Schwierigkeit ist nicht die DNA zu schneiden – dafür gibt es Enzyme, die schon lange bekannt sind – sondern sie an einer definierten Stelle zu schneiden. Das Genomeditierungs-System benötigt also zusätzlich zum Schneidewerkzeug einen Mechanismus, der die gewünschte DNA-Zielsequenz erkennt.
gRNA und Cas9: Erkennen und schneiden
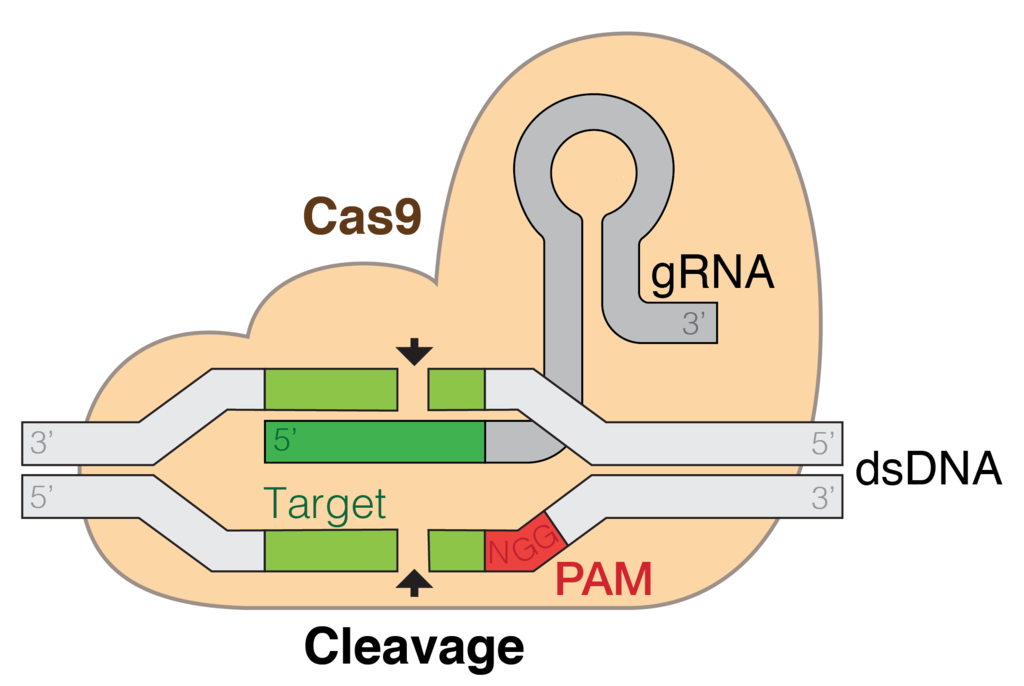
Diese zwei Elemente, die das Fundament der CRISPR/Cas9-Methode bilden, sind die gRNA (guide RNA, was so viel heisst wie führende oder leitende RNA; oft auch single guide RNA genannt) und das Cas9-Protein (CRISPR assoziiertes Protein), die eigentliche "Genschere". Die beiden Elemente bilden zusammen einen Komplex. Die gRNA wird im Labor so synthetisiert, dass sie zur DNA-Zielsequenz komplementär ist – sie erkennt also das Ziel, das geschnitten werden soll. Meist ist sie ca. 17-21 Nukleotide lang; damit wird sichergestellt, dass die Sequenz möglichst nur einmal im Genom vorkommt und es keine Schnitte ausserhalb der gewünschten Region gibt (s. aber den Artikel "CRISPR/Cas: Fragen und Regeln"). Bevor aber der gRNA/Cas9-Komplex die DNA-Sequenz erkennen und schneiden kann, muss er erst mal an sie herankommen.
PAM-Motiv: Sesam öffne dich
PAM-Motive sind nichts anderes als kurze Sequenzen auf der DNA, die von Cas-Proteinen erkannt werden. Im Falle von Cas9, das aus dem Bakterium Streptococcus pyogenes isoliert wurde, handelt es sich dabei um die Sequenz NGG, wobei N jedes Nukleotid sein kann. Das Cas9-Protein bindet an ein PAM-Motiv und entwindet die DNA-Helix an dieser Stelle – nun ist die DNA-Sequenz zugänglich und kann theoretisch geschnitten werden.
Dass die PAM-Motive nur wenige Nukleotide lang sind, ist für die Gentechnik ein Vorteil. Solch kurze Nukleotidsequenzen kommen im Genom relativ häufig vor, so dass meist mehrere PAM innerhalb eines Gens zu finden sind. Die Wahrscheinlichkeit, dass eine PAM in der Nähe der gewünschten Zielsequenz ist, die geschnitten werden soll, ist somit gross. PAM-Motive kommen aber trotzdem nicht immer genau dort vor, wo man die DNA schneiden möchte. Forschende haben aber neben dem Cas9 auch ganz viele andere Cas-Proteine entdeckt, die alle ähnlich funktionieren, aber unterschiedliche PAM-Motive ansteuern. Dies ermöglicht eine flexiblere und noch gezieltere Genomeditierung.